
Les processus mis en jeu lors du développement d'un organisme pluricellulaire ont deux caractéristiques intéressantes à mettre en avant :
Dans cet exemple, nous allons voir comment MGS peut être utilisé pour rendre compte d'un phénomène tel que la neurulation.
Le processus de neurulation consiste en une modification topologique de la région dorsale de l'embryon (cf image ci-dessous) : le plan neural (neural plate) se plie pour former le pli neural (neural fold). Cette déformation courbe alors le feuillet de cellules jusqu'à ce que les cellules jusque là sur des extrémités opposées se touchent pour former le tube neural (neural tube). Finalement, dans une dernière étape, le tube se sépare de l'épiderme (epidermis) au niveau de la gouttière neurale (neural crest). La transformation topologique sous-jacente correspond au passage d'un plan à un tube, ce qui n'est pas trivial à implémenter.
Pour simplifier cet exemple, nous allons considérer un feuillet de cellules épithéliales qui se courbe sous l'effet de migrations et de déformations cellulaires locales. Les cellules qui étaient au début sur des côtés opposés se colleront lorsqu'elles seront assez proches pour former le cylindre.
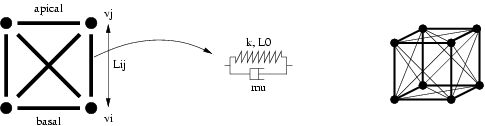
Le modèle mécanique que nous utilisons est inspiré des travaux décrits dans [Odell et al., 1981] et dans [Nagpal, 2001]. Une cellule épithéliale est un cube composé d'un volume pouvant contenir le code génétique, de 6 faces qui présentent une interface d'échange entre cellule (un face commune entre 2 cellules épithéliales décrit la nature des échangent entre ces deux cellules, de 24 arcs (les 12 arcs habituels formant les bords communs des 6 faces, et les 12 diagonales de chaque faces) encore appelés fibres qui simulent le cytosquelette la cellule, et de 8 sommets correspondant aux jonctions des fibres.
Pour ce modèle, nous allons utiliser uniquement les cellules de dimension 0 et 1. En fait, Odell décrit chaque fibre par un ressort en parallèle avec une friction. En modifiant la longueur à vide et/ou le coefficient de raideur du ressort, on peut déformer la cellule.
On définit un type pour chaque cellule :
record Vertex = {Le type Vertex est un enregistrement de 10 champs utilisés pour la mécanique newtonienne : px, py et pz représentent le vecteur position, vx, vy et vz le vecteur vitesse, ax, ay et az le vecteur accélération, et m la masse.
record Edge = {Le type Edge rassemble les différentes constantes associées à la modélisation du ressort k, mu et L0. La vitesse d'élongation de l'arc est stockée dans le champ vL. Enfin, les deux champs vi et vj réfèrent aux bords des arcs ; ils sont utilisés pour générer une pseudo-orientation des arcs.
A l'état initial, on considère une matrice de nxm cellules épithéliales.
Un modèle mécanique est utilisé pour simuler la déformation de la structure du système. Ici, on va calculer le mouvement des sommets qui subissent les forces engendrées par les ressorts. Cela est fait à travers deux transformations :
trans <1> update_spring = {Cette transformation agit sur les éléments de dimension 1 de la collection sur laquelle on l'applique (c'est la signification de trans <1> ). Elle calcule pour chaque arc e la vitesse d'élongation du ressort stockée dans e.vL. On calcule également, sous la forme d'un vecteur, la force appliquée aux extrémités de e.
trans <0> integration [delta_t = 0.01] = {Cette seconde transformation agit sur les sommets. Pour chaque sommet vi , on somme les forces induites par chaque arc incident à vi. On peut remarquer la prise en compte de l'orientation de l'arc par rapport au sommet. On utilise le second principe de la mécanique de Newton pour calculer l'accélération du sommet. On intègre ensuite ce vecteur accélération pour obtenir les vecteurs vitesse et position. L'intégration est faite par la simple méthode d'approximation d'Euler. Le pas de temps delta_t est donné en option à la transformation.
Le film disponible ici (8.4M) permet de visualiser l'effet de cette transformation sur une matrice 20x2 de cellules.
Dans le film précédent, l'issue de la contraction des cellules épithéliales n'est pas celle attendue. En effet, il faut tester la collision entre les bords et coudre ces bords pour former le cylindre. Cette couture peut être représentée graphiquement de la façon suivante :
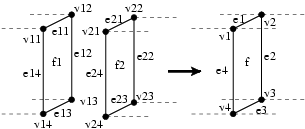
La transformation est alors définie ainsi :
patch surgery = {Le filtre résume la figure présentée ci-dessus. Le prédicat P vérifie les faces f1 et f2 sont globalement confondues, ce qui est synonyme de collision. En partie droite, on reconstruit une face correspondant à la fusion de f1 et f2. Les fonctions average_i(a,b) fait la moyenne des valeurs associées aux deux i-cellules a et b. La fonction unmatched_cofaces retourne la liste des cofaces communes des éléments passés en paramètres à l'exception des éléments déjà filtrés.
Les captures d'écran ci-dessous présentent la chirurgie topologique :
 |
 |
 |
Une animation est disponible ici (6.6M).
Pour vérifier que la couture ferme bel et bien le cylindre, nous allons insérer dans les cellules de dimension 3 un jeton qui va diffuser de cellule en cellule. Lorsqu'il est sur bord, il ne peut évidemment pas sortir du feuillet. Or après couture, les bords n'existent plus et le jeton peut passer. On suppose pour cela qu'il n'y a qu'une seule rangée de cellule ; il s'agit donc d'une séquence de cellules qui se courbe pour former un anneau. La transformation suivante réalise la diffusion du dit jeton :
trans <3,2> token = {A l'état initial tous les volumes ont la valeur `cell, exceptés deux cellules consécutives dont l'une est marquée par `token (elle contient le jeton) et l'autre par `back. La marque `back oblige le jeton à se déplacer dans une seule direction. En effet, si on observe la première règle, le jeton `token prend la place d'une cellule vide et le jeton `back le suit de près. Les deux jetons échangent leur position dans la deuxième règle lorsqu'ils se situent à l'extrémité de la séquence.
Une animation est disponible ici (3.6M).
[Odell et al., 1981] Odell, G.-M., Oster, G., Alberch, P., and Burnside, B. (1981). The mechanical basis of morphogenesis. i. epithelial folding and invagination. Developemental Biology, 85(2):446 462.
[Nagpal, 2001] Nagpal, R. (2001). Programmable Self-Assembly: Constructing Global Shape using Biologically-inspired Local Interactions and Origami Mathematics. PhD thesis, Massachusetts Institute of Technology.
| Back
to top |
MGS examples index |
MGS home page |

Pictures, graphics and animations are licensed under a Creative Commons License.
|
|